In questa nuova esperienza abbiamo calcolato la concentrazione delle proteine nei nostri campioni, precedentemente preparati nell'esperienza di estrazione delle proteine dal lievito.
In questa nuova esperienza abbiamo calcolato la concentrazione delle proteine nei nostri campioni, precedentemente preparati nell'esperienza di estrazione delle proteine dal lievito. Il metodo da noi usato prevede l'utilizzo del reattivo di Bradford (Coomassie Brillant Blue G250, preparato in precedenza) e dello spettrofotometro.
Per iniziare abbiamo fatto una retta di taratura a concentrazioni note, quindi abbiamo perparato un bianco, e 4 standard (25, 50, 75, 100 µg)
Preparazione del bianco
- 0,100 ml acqua
- 5 ml di reattivo di Bradford
Preparazione dei campioni
| STD 25 µg | STD 50 µg | STD 75 µg | STD 100 µg | |
| BSA 1mg/ml | 0,025 ml | 0,050 ml | 0,075 ml | 0,100 ml |
| Acqua | 0,075 ml | 0,050 ml | 0,025 ml | |
| Reattivo di Bradford | 5 ml | 5 ml | 5 ml | 5 ml |
Preparazione del campione

- 0,090 ml di acqua
- 0,010 ml di campione
- 5 ml di reattivo di Bradford
Agitare, aspettare 5 minuti e poi effettuare la lettura allo spettrofotometro a lunghezza d'onda 595 nm
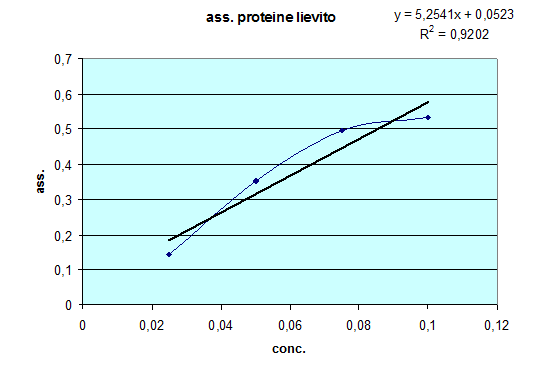
Una volta ottenuti i risultati si disegna una rette di taratura con gli standard.
| Assorbanza | Concentrazioni | ||
| 0,14254 | 0,025 | ||
| 0,35221 | 0,05 | ||
| 0,4952 | 0,075 | ||
| 0,53272 | 0,1 | ||
| Ass. incognite | Conc. incognite | ||
| 0,1956 | 0,027273938 | conc. Roby | |
| 0,24432 | 0,036546697 | conc. Sara |
Trovata la concentrazione che sta in 10 ml del campione si trova quante proteine ci sono in un millilitro.
10 ml : 36,54 µ = 1 ml : x
x = (36,54 µg * 1 µl) / 10 µl = 3,654 µg/µl
Autori: Roberta e Sara